ChIP-seq技術(shù)簡介
染色質(zhì)免疫沉淀后測序(ChIP seq)是一種針對DNA結(jié)合蛋白、組蛋白修飾或核小體的全基因組分析技術(shù)。由于二代測序技術(shù)的巨大進(jìn)步,ChIP-seq比其最初版本ChIP-chip具有更高的分辨率、更低的噪聲和更大的覆蓋范圍。隨著測序成本的降低,ChIP- seq已成為研究基因調(diào)控和表觀遺傳機(jī)制不可或缺的工具。
1. 原理
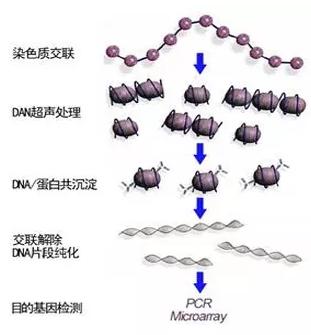
甲醛處理細(xì)胞使目標(biāo)蛋白與DNA交聯(lián),通過超聲波將交聯(lián)后的染色質(zhì)打斷成小片段,一般在200-600bp范圍內(nèi)。再利用抗原抗體的特異性識別反應(yīng),將與目的蛋白相結(jié)合的DNA片段沉淀下來。最后,去交聯(lián)并對純化后DNA進(jìn)行PCR擴(kuò)增,高通量測序,最后與已有基因組序列進(jìn)行比對,以確定 DNA與蛋白質(zhì)結(jié)合的序列。
2.問題分析
2.1.甲醛交聯(lián)對后續(xù)結(jié)果分析的影響?
自從Orlando V等人首次發(fā)明了ChIP技術(shù),十幾年后核心步驟仍無大變化。然而,ChIP-seq技術(shù)實(shí)際存在一定的缺陷,例如甲醛交聯(lián)。甲醛雖然是一種高度滲透的交聯(lián)劑,但由于其反應(yīng)活性僅限于胺,因此其交聯(lián)效率較低;對哺乳動(dòng)物細(xì)胞而言,其最大交聯(lián)效率僅為1%。因此甲醛交聯(lián)細(xì)胞所需的起始量很大,也因此該技術(shù)也很難適用于微量細(xì)胞及單細(xì)胞樣本。牛津大學(xué)出版社發(fā)表的文章In vivo formaldehyde cross-linking: it is time for black box analysis指出,某些蛋白質(zhì)如:阻遏蛋白,NF-κB等無法通過甲醛交聯(lián)到DNA上,研究表明;在DNA上的停留時(shí)間短于5秒的蛋白質(zhì)無法用甲醛交聯(lián)。其次,甲醛會(huì)導(dǎo)致許多其它無關(guān)蛋白質(zhì)交聯(lián)到DNA上,影響后續(xù)分析數(shù)據(jù)。有報(bào)道稱,甲醛交聯(lián)會(huì)觸發(fā)DNA損傷應(yīng)答機(jī)制,從而改變?nèi)旧w組分,進(jìn)而使ChIP結(jié)果產(chǎn)生偏向性。除此之外,由于交聯(lián)反應(yīng)在加熱和低PH的情況下會(huì)發(fā)生逆轉(zhuǎn),因此DNA蛋與白質(zhì)的交聯(lián)復(fù)合物的穩(wěn)定性也是一個(gè)值得關(guān)注的問題。因此,甲醛究竟在多大程度上能反應(yīng)細(xì)胞內(nèi)蛋白質(zhì)的分布仍不能確定。
根據(jù)有無甲醛交聯(lián)步驟可以把CHIP技術(shù)劃分為兩種類型,一類是存在甲醛交聯(lián)的 ChIP,即X-ChIP(cross-linking and mechanical shearing ChIP);另一類是無交聯(lián)存在的ChIP,即N-ChIP(native-ChIP);相較于X-ChIP,N-ChIP技術(shù)有一系列的優(yōu)點(diǎn),包括:高分辨率(MNase的使用使得染色體的片段化可以小至核小體)、避免了甲醛交聯(lián)帶來的非特異性蛋白在DNA上的富集、避免了甲醛交聯(lián)對抗原表位的遮蓋、步驟的減少降低了樣品的損失等。然而,由于使用了MNase,N-ChIP只適用于研究組蛋白修飾,大部分情況下不能用于轉(zhuǎn)錄因子的研究。
2.2. 超聲波打斷和酶斷裂方法的比較?
酶類:最常用的酶類如MNase,即:微球菌核酸酶,是一種能降解核小體連接區(qū)的DNA序列的核酸酶,最初從金黃色葡萄球菌中分離出來。MNase消化染色質(zhì)可以釋放出一個(gè)個(gè)獨(dú)立的核小體。
MNase酶解法具有一定的局限性,首先,MNase對于偏向于切割A(yù)/T堿基位點(diǎn),導(dǎo)致核小體在A/T富集區(qū)域的表達(dá)量低于真實(shí)情況;其次,MNase不能在核小體邊界精確切割,這導(dǎo)致在確定染色質(zhì)的開放位置與真實(shí)情況存在差異;而且,MNase偏向于消化脆性核小體。在不同物種的實(shí)驗(yàn)證據(jù)表明,脆性核小體占據(jù)了基因啟動(dòng)子和轉(zhuǎn)錄終止位點(diǎn),而脆性核小體只在MNase濃度較低且消化時(shí)間較短的情況下才能被檢測出來,因此很難將脆弱核小體量化到穩(wěn)定核小體的相對豐度。
超聲打斷則不如酶裂解法溫和,而且由于打斷的不均勻性,導(dǎo)致測序結(jié)果背景噪音高,影響后續(xù)數(shù)據(jù)分析。由于文章篇幅限制,在此不多贅述。
那么究竟選擇酶解還是超聲打斷,需要視情況而定。可參考以下建議:
如果所研究的蛋白質(zhì)高豐度表達(dá)且與DNA結(jié)合緊密如組蛋白,那么樣本無需需交聯(lián),這時(shí)可使用酶解法。
如果所研究的蛋白質(zhì)表達(dá)豐度較低或與DNA結(jié)合不緊密如轉(zhuǎn)錄因子等,往往需要用交聯(lián)試劑將樣本進(jìn)行固定,穩(wěn)定蛋白質(zhì)和DNA的形態(tài),這時(shí)最好選用超聲法進(jìn)行斷裂。
3.拓展
3.1適用于微量細(xì)胞水平的ChIP技術(shù)及其原理
(1)CUT-Tag技術(shù)
CUT-Tag可同時(shí)用于研究轉(zhuǎn)錄因子結(jié)合位點(diǎn)以及DNA的開放性。可在一天之內(nèi)完成從細(xì)胞到建庫完成的所有步驟,且具有高分辨率,低噪音等特點(diǎn)。起始細(xì)胞用量可低至50個(gè)。
原理
利用抗原抗體特異性反應(yīng),加入特異性抗體與染色體上目標(biāo)蛋白結(jié)合,加入二抗與該抗體結(jié)合以募集更多的pA-Tn5轉(zhuǎn)座酶復(fù)合物至目標(biāo)蛋白與DNA序列的結(jié)合位點(diǎn)上,轉(zhuǎn)座酶復(fù)合物切割該染色質(zhì)開放位點(diǎn),并在切割后的DNA片段兩端加入接頭,文庫構(gòu)建完成,可直接進(jìn)行后續(xù)測序,與已有基因組序列比對,即可知道目標(biāo)蛋白與DNA的結(jié)合位點(diǎn)。

(2)ChIL-seq
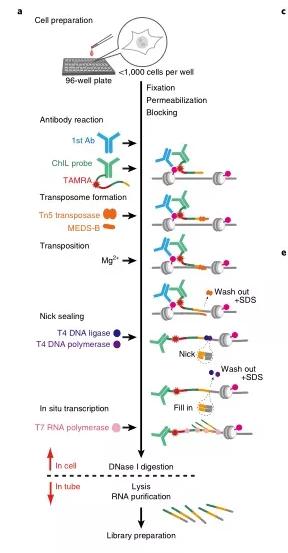
ChIL(chromatin integration labelling),即染色質(zhì)集成標(biāo)簽技術(shù),可同時(shí)用于研究轉(zhuǎn)錄因子結(jié)合位點(diǎn)以及DNA的開放性,起始細(xì)胞用量為100-1000個(gè)細(xì)胞。ChIL-seq 避免了傳統(tǒng)ChIP-seq技術(shù)中由于抗體沉淀所帶來的回收率低的缺陷,尤其適用于貼壁細(xì)胞。對于活躍的組蛋白標(biāo)記如H3K4me3 ,H3K27ac,起始細(xì)胞用量甚至可降低至單細(xì)胞水平。
原理
在96孔板中加入細(xì)胞,經(jīng)固定,染色后加入一抗與目標(biāo)蛋白結(jié)合,隨后加入ChIL探針(由二抗和ChIL DNA組成),探針中的ChIL DNA經(jīng)Tn5轉(zhuǎn)座酶整合到目標(biāo)蛋白所在基因組DNA附近,隨后T7 RNA 聚合酶經(jīng)ChIL DNA中的啟動(dòng)子啟動(dòng)轉(zhuǎn)錄,以此處基因組DNA為模板合成RNA,經(jīng)DNase I消化和裂解釋放RNA,以純化的RNA建庫測序。
(3)Drop-ChIP
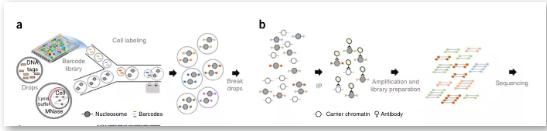
Drop-ChIP:使用了特定的微流控裝置,分辨率可達(dá)到單細(xì)胞水平。該技術(shù)不僅可以在單細(xì)胞水平研究轉(zhuǎn)錄因子結(jié)合位點(diǎn)及組蛋白修飾,還可以從細(xì)胞特異性角度研究不同細(xì)胞間染色質(zhì)的變異程度。我們認(rèn)為,整合單細(xì)胞染色質(zhì)和單細(xì)胞表達(dá)數(shù)據(jù),可以使調(diào)控元件與靶基因更精確地耦合,并更深入地了解它們的功能動(dòng)力學(xué)和關(guān)系。
原理
首先,將待研究的細(xì)胞與裂解液,MNase混合進(jìn)行染色質(zhì)消化,另外設(shè)計(jì)一個(gè)包含很多種不同接頭的液滴,在微流控裝置上反應(yīng),使得每一個(gè)細(xì)胞液滴與一種接頭液滴混合,同時(shí)與含有DNA連接酶的buffer液滴混合。這個(gè)過程中,接頭序列自動(dòng)連接到裂解的染色質(zhì)片段兩端,進(jìn)而進(jìn)行細(xì)胞裂解,使用特異性抗體對目標(biāo)蛋白進(jìn)行沉淀,對富集后的DNA進(jìn)行測序。與已有基因組序列比對即可知道目標(biāo)蛋白作用位點(diǎn)。
參考文獻(xiàn):
[1]. 1: Kaya-Okur HS, Wu SJ, Codomo CA, Pledger ES, Bryson TD, Henikoff JG, Ahmad K,Henikoff S. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019 Apr 29;10(1):1930.
[2]. Harada A, Maehara K, Handa T, Arimura Y, Nogami J, Hayashi-Takanaka Y,
Shirahige K, Kurumizaka H, Kimura H, Ohkawa Y. A chromatin integration labelling method enables epigenomic profiling with lower input. Nat Cell Biol. 2019 Feb;21(2):287-296.
[3]. Rotem A, Ram O, Shoresh N, Sperling RA, Goren A, Weitz DA, Bernstein BE.
Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nat Biotechnol. 2015 Nov;33(11):1165-72
免責(zé)聲明:文章來源-易基因(微信公眾號) 作者:e-gene,以傳播知識、有益學(xué)習(xí)和研究為宗旨, 如內(nèi)容有誤,請批評指正。 轉(zhuǎn)載僅供參考學(xué)習(xí)及傳遞有用信息,版權(quán)歸原作者所有,如侵犯權(quán)益,請聯(lián)系刪除。